山西农业大学农业基因资源研究中心联合华大科技在Scientific Data(JCR Q1,IF5year=8.9)上发表题为“A complete reference genome of broomcorn millet”的文章。该研究成功构建黍稷(AJ8)端粒到端粒(T2T)Gap-Free参考基因组,为黍稷这种重要谷类作物的种群及保护遗传研究和育种提供了宝贵的遗传资源。

一、基因组组装与组装质量评估
研究团队利用77 Gb PacBio HiFi数据(89.3X,N50=17,996 bp)、165.6 Gb Oxford Nanopore超长(196X,N50=55,765 bp)和252.98 Gb高通量染色体构象捕获(Hi-C)DNBSEQ PE150测序数据(293.5X),成功构建黍稷(AJ8)端粒到端粒(T2T)Gap-Free参考基因组。
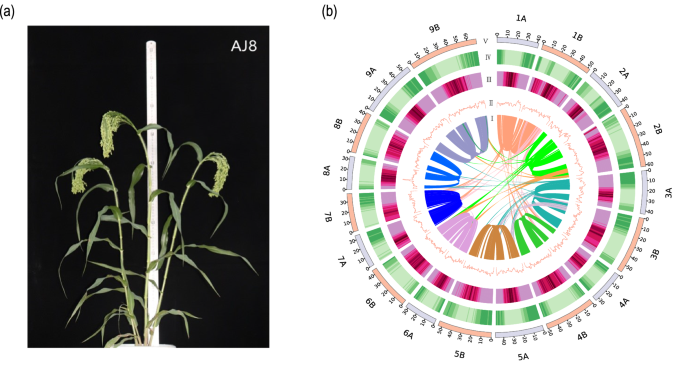
图1 (a) AJ8植物照片
(b) AJ8基因组的基因组Circos图
组装得到的AJ8基因组大小约为834.7 Mb,Contig N50=48.3 Mb,包含18条染色体,并识别了18个着丝粒和36个端粒。且在完整性方面表现出较高质量:BUSCO评分99.6%,QV:61.7,LTR组装指数(LAI)值:20.4。
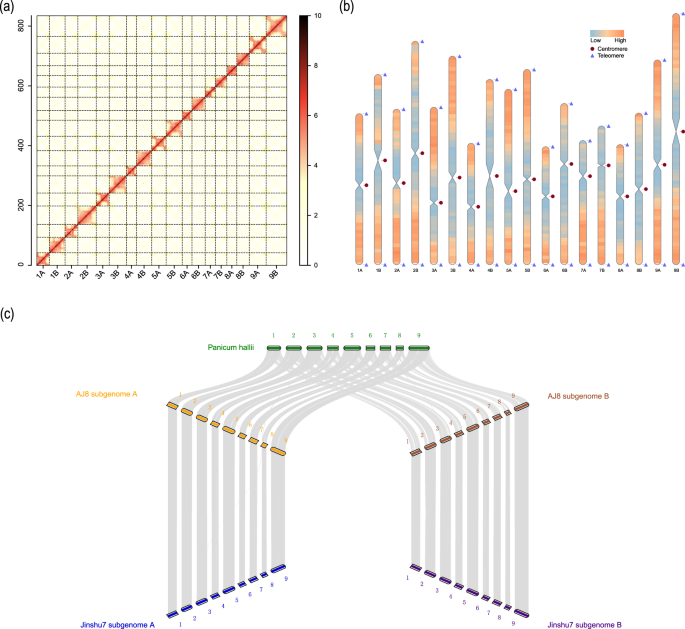
图2 AJ8基因组组装质量评估
(a) Hi-C热图 (b)端粒和着丝粒检测
(c) AJ8与晋黍7号(Jinshu7)、黍稷二倍体近缘物种Panicum hallii基因组同源性分析
二、基因组注释
研究使用RepeatModeler(v1.0.4)和LTR-FINDER(v1.0.7)构建de novo重复序列库用于检测散在重复和低复杂度序列,随后使用RepeatMasker(v4.0.7),RepeatProteinMasker(v4.0.7)识别DNA和蛋白质转座因子(TE)。利用TandemRepeatFinder(v4.10.0)检测串联重复,共获得433.8 Mb(~52.0%)重复序列。
采用基于转录组预测、同源比对预测、从头预测的方法进行基因预测和功能注释,共鉴定到63,678个基因。
研究将AJ8与其他物种基因组的基因长度分布进行比较,结果表明,AJ8与玉米、高粱、陇糜4号(LM_v1)、晋黍7号(Jinshu7)呈相似的基因长度分布模式(图3a)。BUSCO分析表明,在1,614个胚性植物单拷贝直系同源物中,99.5%(单拷贝基因:19.5%,重复基因:80.0%)被成功鉴定为完整的,而0.2%的基因在组装过程中出现碎片,0.3%的基因缺失(图3b)。62,885个(98.8%)基因在多个数据库中被成功注释,51,958个基因(81.6%)在21个RNA-seq测序样本中表现出转录活性(FPKM值≥1)(图3c,图3d)。
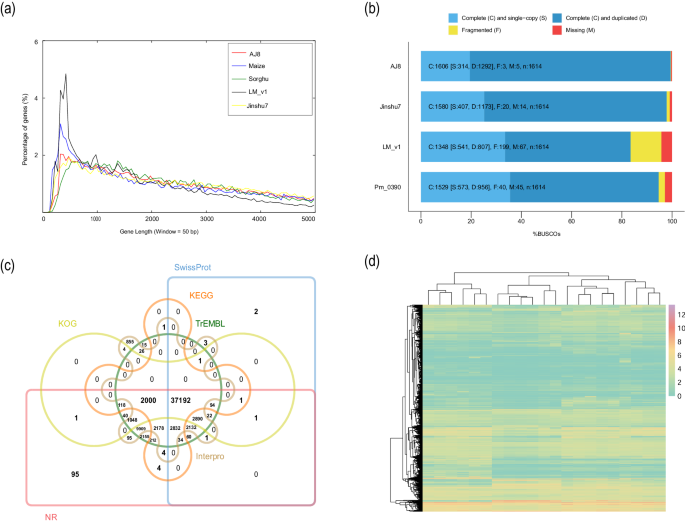
图3基因预测质量评估
黍稷T2T Gap-Free完整基因组的完成,为揭示AJ8的抗旱和适应机制提供了坚实的基础,并且为黍稷及其他谷类作物的种群及保护遗传研究和快速育种提供了重要的遗传资源参考。
山西农业大学农业基因资源研究中心王海岗、王君杰,华大科技陈春海等为论文共同第一作者,山西农业大学农业基因资源研究中心穆志新、王纶、乔治军为论文共同通讯作者。该研究得到山西省重点研发计划、山西省农业种质资源保护与利用项目、中央引导地方科技发展资金和国家现代农业产业技术体系建设专项资助。


